王二涛研究组及合作团队提出根际微生物群落“扩增-选择”组装新模型
2020年3月7日,Science Bulletin在线发表中国科学院分子植物科学卓越创新中心王二涛研究组及其合作团队完成的题为“An amplification-selection model for quantified rhizosphere microbiota assembly”的研究论文,该研究基于微生物绝对丰度提出了植物根际微生物群落“扩增-选择”组装的新模型。
在自然界中,植物根系与大量的微生物互作,这些微生物定殖在根际土中,或附着于根系表面,或定殖于根内,统称为植物根际微生物组。植物根际微生物对于植物的生长发育和环境适应具有重要作用。基于传统的微生物相对丰度(the relative abundance,通过高通量测序16S/18S rRNA基因获得环境样本中各微生物群落的相对组成)数据,研究人员提出了根际微生物群落的两步或多步筛选组装模型(two-step selection model 或者multi-step selection model),该模型认为:微生物依次在根外土(bulk soil)、根际土 (rhizosphere soil) 和根内 (root) 逐步被筛选,形成植物根际特异的微生物群落(图示)。其中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)是根际富集的主要菌群,而酸酐菌门(Acidobacteria)是根际排斥的主要菌群(图示)。
基于微生物相对丰度的研究描述了不同植物物种根际微生物的群落组成及多样性,但忽略了单位质量或体积中不同微生物群落的绝对丰度(the absolute abundance)。另外,基于16S/18S rRNA基因的相对丰度研究忽视了不同细菌/真菌的16S/18S rRNA基因的拷贝数差异。因此,相对丰度的研究结果很难与定量的植物生理生态指标进行联系,阻碍了我们对于根际微生物生态功能的理解。
通过定量蒺藜苜蓿的根际微生物组,我们发现平均每克根外土、根际土和根系中细菌16S rRNA基因的含量为1.1 × 109,1.51 × 1010和3.28 × 109,根际土中细菌16S rRNA基因的含量是根外土的13.7倍。当对16S rRNA基因的拷贝数进行加权后,我们发现平均每克根外土、根际土和根系中细菌数目为6.91 × 108,6.44 × 109和1.24 × 109,根际土中细菌细胞数目是根外土的9.3倍。表明相对于根外土,细菌的绝对丰度在根际土中是显著升高的。进一步分析根内微生物群落相对于根际微生物群落的比例,我们发现放线菌门、γ-变形菌门及Δ-变形菌门从根际土进入根内的比例最大,分别为63%、59%和59%。
基于这些研究结果,我们提出根际微生物群落组装“扩增-选择”的新模型,该模型认为,与根外土相比,主要菌门在根际土中的绝对丰度都被显著扩增,经根际土扩增的微生物进一步被根筛选,形成特异的根内微生物群落。我们把根外土比作“乡村”或者“沙漠”,把根际土比作“大都市”或者“绿洲”,相对于营养较为贫乏的“乡村”,“大都市”可以为微生物提供更多的工作机会,大部分微生物能在“大都市”找到岗位并繁衍扩增。不同微生物扩增倍数可能由微生物自身扩增速率,微生物与微生物互作,植物与微生物互作等因素决定。根际微生物群落组装的“扩增-选择”新模型,将指导我们定量追踪植物根际微生物群在不同生长周期绝对丰度的变化,有助于将根际微生物与植物定量性状关联起来,以便更好利用根际微生物提升农业的可持续发展。
该工作是中科院分子植物科学卓越创新中心王二涛研究组与上海师范大学于楠研究组和河南大学张学斌研究组合作完成的,王孝林博士为第一作者。王二涛研究员,于楠副教授和张学斌教授为论文共同通讯作者。该研究受中国科学院和国家自然基金委的资助。
论文链接:https://doi.org/10.1016/j.scib.2020.03.005
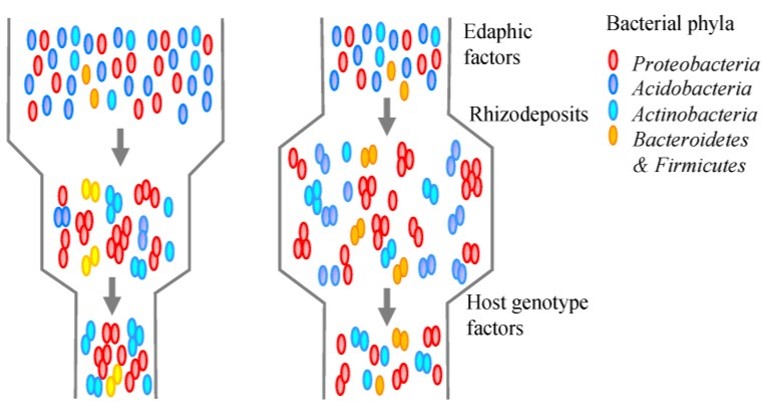
根际微生物群落“扩增-选择”组装新模型:左图为基于微生物相对量的根际微生物群落“两步选择”模型(two-step selection model),右图为根际微生物群落“扩增-选择”组装新模型(amplification-selection model)。