Jungnam Cho研究组建立了一种检测转座子中间产物的新方法
2018年12月11日,《Nature Plants》杂志在线发表了中科院分子植物科学卓越创新中心/植物生理生态研究所,同时隶属中国科学院-英国约翰.英纳斯中心联合项目(Center of Excellence for Plant and Microbial Sciences; CEPAMS)JungnamCho研究组题为“Sensitivedetection ofpre-integration intermediates of LTR retrotransposons in cropplants”的研究论文,本文提出了一种可以灵敏检测农作物(如水稻)中的反转录转座子转座过程中所产生的中间产物的新方法。
反转录转座子在宿主基因组进化过程中扮演着重要角色。研究者主要从它们在进化过程中所保留的DNA序列信息中推测其重要性,但这样的研究只能推测转座子历史进化过程中发生的事件,却不能预测其现在所拥有的转座潜能。之前有研究提出了一种名为Sequence-Independent Retrotransposon Trapping (SIRT)的技术用来确定染色体外线性DNA(extrachromosomal linear DNA ,eclDNA)的存在,以此来提示拟南芥中活跃的LTR反转录转座子的存在。然而,SIRT不能用于像水稻那种庞大且转座子富集的基因组。本研究提出了一种名为amplification of LTR of eclDNAs followed by sequencing(ALE-seq)的新方法来弥补SIRT的不足。ALE-seq在经过体外的转录和反转录过程后对LTR 5’末端进行测序。利用这个方法,本研究检测到一个属于Copia家族,且在高温条件下会被激活的LTR反转录转座子Go-on的eclDNA。与此同时,我们在对不同的水稻材料进行测序时发现,在高温条件下,Go-on优先在籼稻中积累。另外,将ALE-seq用于番茄中确定了在果实发育过程中有调节作用的Gypsy家族的反转录转座子。
这是一个适用于ALE-seq分析的生物信息学的传输途径,它可以直接用于发现新的活跃的反转录转座子,并且不需要参考注释信息。因此,这个途径有利于对那些没有参考基因组信息或者参考基因组信息质量很低的机体的LTR反转录转座子活性进行评估。
论文链接:http://dx.doi.org/10.1038/s41477-018-0320-9
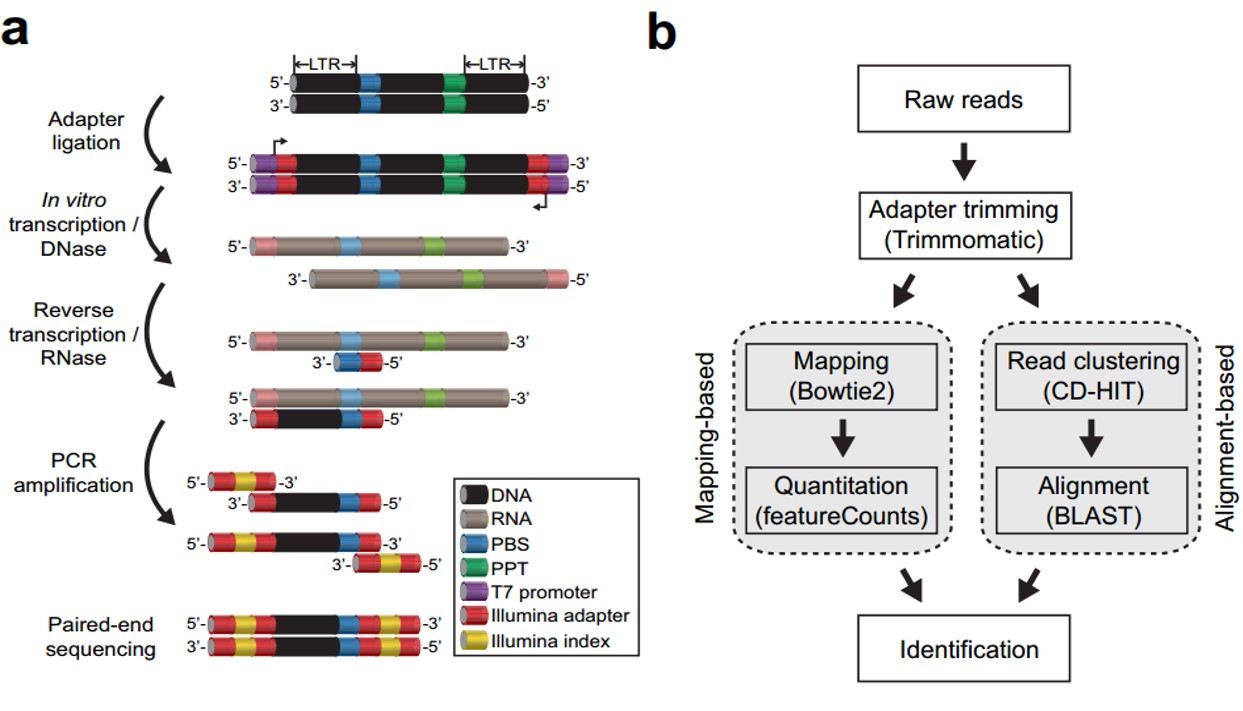
The workflow and Analysis pipelineof ALE-seq.