李轩研究组通过组学大数据研究揭示了RNA编辑表观遗传位点的系统进化规律
7月28日,《PLOS Genetics》杂志发表了我所李轩研究组题为“The landscape of A-to-I RNA editome is shaped by both positive and purifying selection”的研究论文。 该工作通过对多生物物种RNA编辑事件的系统发现和分析,首次揭示了RNA编辑表观遗传位点的系统进化规律,及其主要在动物神经功能和神经发育上的作用。
RNA编辑是转录后RNA分子发生碱基插入、删除、或替换,从而导致基因编码序列改变的机制。目前研究最多的是A-to-I RNA编辑,即广泛存在于后生动物中RNA上腺嘌呤(A)脱氨基变为次黄嘌呤(I)的碱基变换。A-to-I编辑是由一类作用于RNA分子的腺苷脱氨酶(ADAR)催化,主要发生在RNA双链结构区域。而次黄嘌呤在翻译的过程中会被识别成为鸟嘌呤(G),带来mRNA 编码改变,增加遗传多样性。针对A-to-I RNA编辑的表观遗传作用,存在一系列关键问题亟待回答。 首先, A-to-I RNA编辑是如何、并在多大程度上影响转录组编码基因的多样性? 其次,RNA编辑事件本身在进化中如何被约束和选择? RNA编辑的表观遗传作用是否导致相关基因的进化动力学改变? 最后,RNA编辑的表观遗传作用带给宿主的适应性是通过哪些功能怎样实现的?进化中被选择的有益RNA编辑事件,有怎样的功能偏好性?
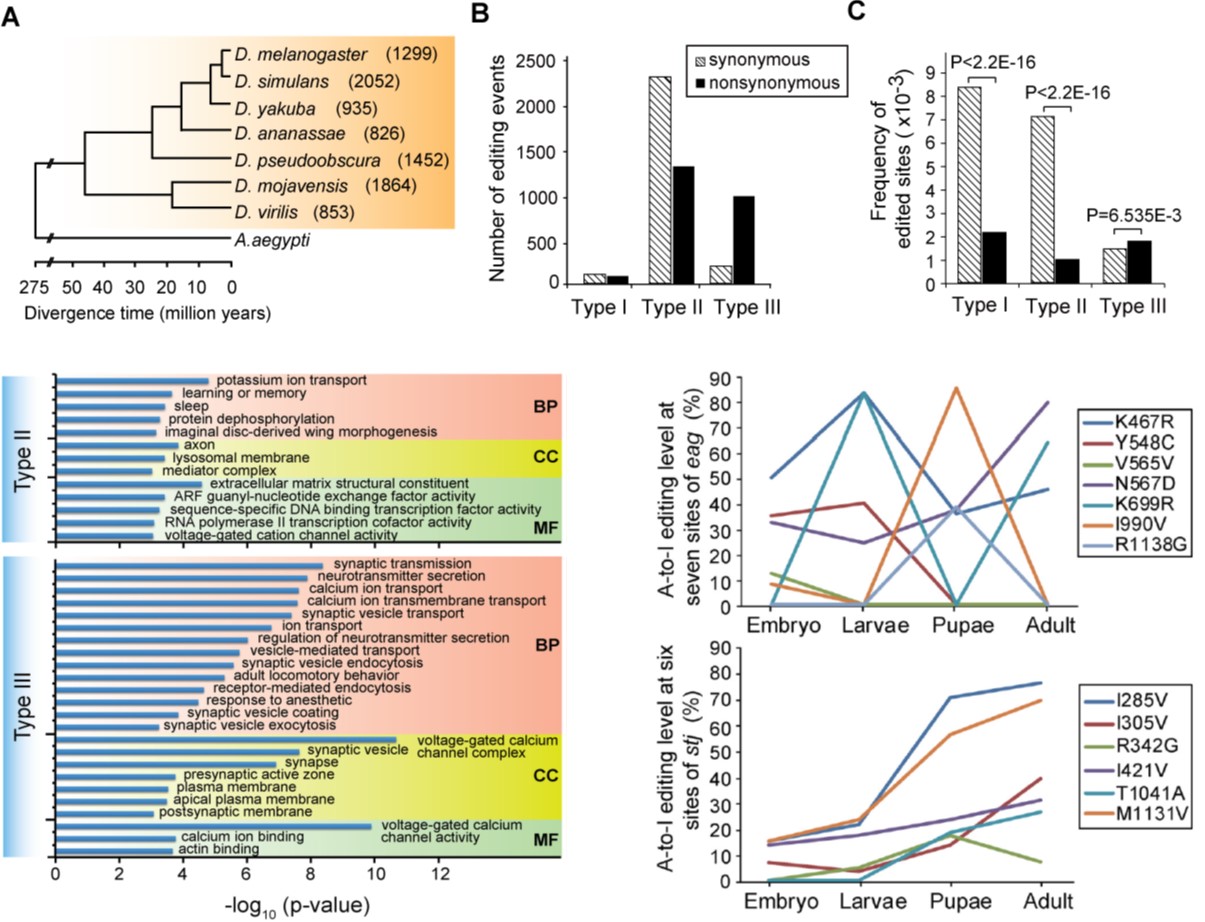
针对这些问题,李轩研究组博士生周红霞、博士后余曜等人,与巴斯德研究所郝沛研究员、美国密之根大学王红兵教授合作,利用果蝇7个物种构建了一个由进化上相近的物种(进化距离~4500万年)组成的模型系统,并开展特定时间跨度内A-I RNA 编辑事件的发现和进化追踪。通过收集多维转录组大数据、系统筛选和鉴定,该研究共发现了9281个A-to-I RNA编辑事件,其中5150 (55.5%) 分布于2734个基因的编码区(CDS)。进化遗传分析发现这2734 个基因属于1526个同源基因家族,约占7个果蝇物种总基因家族的5%。根据CDS区内编辑位点的保守性,这5150编辑事件被分成3种不同的类型:第一类位点发生在单基因家族基因上;第二类发生在多基因家族基因上,但位点不保守;第三类发生在多基因家族基因上,且位点保守。对这三类位点及其基因进行选择分析发现,第一和第二类位点均受到纯化选择(负选择)影响,而只有第三类位点受到正选择压力。重要的是,发现第三类位点高度富集于神经系统的元件和功能中。通过对这三类编辑位点进行不同组织、不同发育时期以及动物变态发育过程中的分布及变化分析,第一次发现了A-to-I RNA编辑在动物发育、交配(mating)等生理过程中动态变化的证据,进一步支持了三类不同编辑位点的重要功能。这些结果都指向神经系统功能,说明了RNA编辑表观遗传作用的适应性主要通过神经系统功能实现。神经系统功能是检验有益RNA编辑位点主要标准。以上发现,揭示了由RNA编辑表观遗传机制引入的编码可塑性,而产生一类新的二分变异。在二倍体有性生殖系统中,它是维持基因表达杂合性的一个重要机制,对克服等位杂合子分离有不可替代的优势。
该工作得到了国家重点基础研究(973计划)和自然科学基金委项目的支持。
文章链接http://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1006191